“为何先进的基因检测仍找不出病因?”临床实践中,医生可能会遭遇到患者或家属发出的这类质疑。特别是当部分患者的基因检测报告可能显示“阴性”结果,或检测结果难以完全解释患者临床症状时,质疑声会越发高涨。这类患者的病因,可能与基因组中一个长期以来研究相对有限的基因组“暗区”——非编码区有关。
如果基因是一本书,外显子就是书中直接传达故事情节的文字,而非编码区则是段落间的空白。传统观念认为,只有“文字”才承载信息。然而,现代遗传学发现,这些“空白”区域同样藏着关键指令,一旦出错也会导致疾病发生。
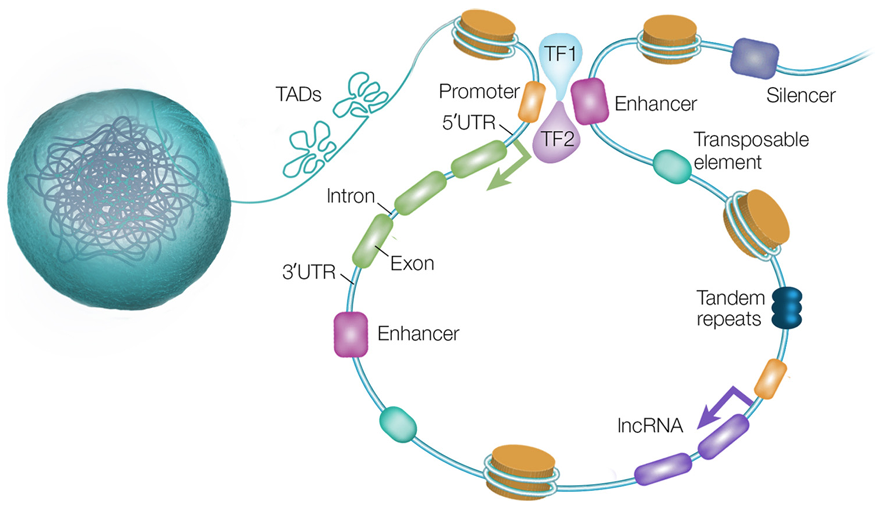
人类非编码基因组中的功能元件[1]
被忽视的基因组“暗区”:非编码区
在人类基因组中,编码蛋白质的外显子区域仅占约2%,而非编码区域占据了98%。20世纪末至21世纪初,部分研究曾将非编码区称为“垃圾DNA”,但实际情况并非如此。
非编码区主要包括内含子、调控元件(如启动子、增强子)、非编码RNA基因及染色体结构区域(如端粒、着丝粒)等。在遗传病致病变异研究中,除经典的外显子变异及近剪接位点变异(指距离外显子-内含子边界±2bp内的变异)外,越来越多研究发现了致病的深度内含子变异(指距离剪接位点50个碱基以上的DNA序列改变)。这类变异可能通过多种机制致病:
► 创造新的剪接位点,导致异常“假外显子”插入成熟mRNA;
► 破坏正常的剪接增强子或沉默子;
► 影响基因的调控元件;
► 改变非编码RNA的功能。
典型案例:破解苯丙酮尿症家庭的“第二变异”之谜
一对夫妇曾生育一个苯丙酮尿症患儿(现年7岁)。该患儿既往在外院经全外显子组测序(WES),仅检出母源性致病变异(位于PAH基因11号外显子区域),但始终未能明确第二致病等位基因。
为了评估家族再生育风险,患者接受了迪安诊断增强型全外显子组测序技术(扩展覆盖深度内含子区域)复测。除了再次确认患儿携带母源PAH基因11号外显子区域的致病性杂合变异以外,突破性检出了父源性深度内含子变异(位于PAH基因10号内含子区域),明确了该患儿病因,为夫妇再生育提供精准遗传咨询指导。

全外显子组测序技术(扩展覆盖深度内含子区域)检测结果
传统检测的盲区:为什么WES或靶向基因Panel,会遗漏这些变异?
临床常用的全外显子组测序 (WES) 或者靶向基因Panel,主要针对外显子编码区域设计,其技术原理决定了它在检测非编码区变异方面存在天然局限:
# 捕获探针覆盖不足
常规WES探针主要靶向外显子编码区及邻近剪接位点,对深度内含子区域、启动子、增强子等非编码区域捕获效率极低。
# 同聚物区域测序偏差
如腺嘌呤、鸟嘌呤等连续重复的同聚物区域易出现测序错误。
# 结构变异识别困难
大片段缺失/插入的断点常位于非编码区,WES因短读长特性无法跨越断点区域,难以精确定位。
#注释信息缺乏
现有数据库中收录的变异,集中于编码区,对非编码区变异的功能注释不足,限制了非编码区致病变异的解读。
如何照亮基因组“暗区”?
面对非编码区致病变异对临床诊断带来的挑战,我们可以采取多层次的解决方案:
1 扩展捕获探针
在WES检测探针中针对性补充已知致病非编码区的捕获探针,提升基因组“暗区”变异的检出率。
2 RNA-seq补充
RNA-seq需结合DNA测序结果,重点分析异常剪接事件或差异表达基因。
3 WGS策略
对临床高度怀疑单基因病,但WES检测结果为阴性的患者,可补充全基因组测序 (WGS) 策略。
4 功能验证
对WES或WGS检出的可能影响剪接的变异,可进行minigene剪接实验等功能实验验证。
随着基因组学技术的进步,我们正逐步揭开基因组“暗区”的神秘面纱。非编码区不再是理论上的概念,而是具有实实在在临床诊断价值的区域。对这些区域的拓展检测,可能是解开疑难病例诊断之谜的关键密码,将为疑难未确诊患者带来新的希望。
迪安诊断采用创新性的探针设计思路,将非编码区已知致病性变异补充进多项遗传病检测探针中,助力遗传病的精准诊断和个性化健康管理。
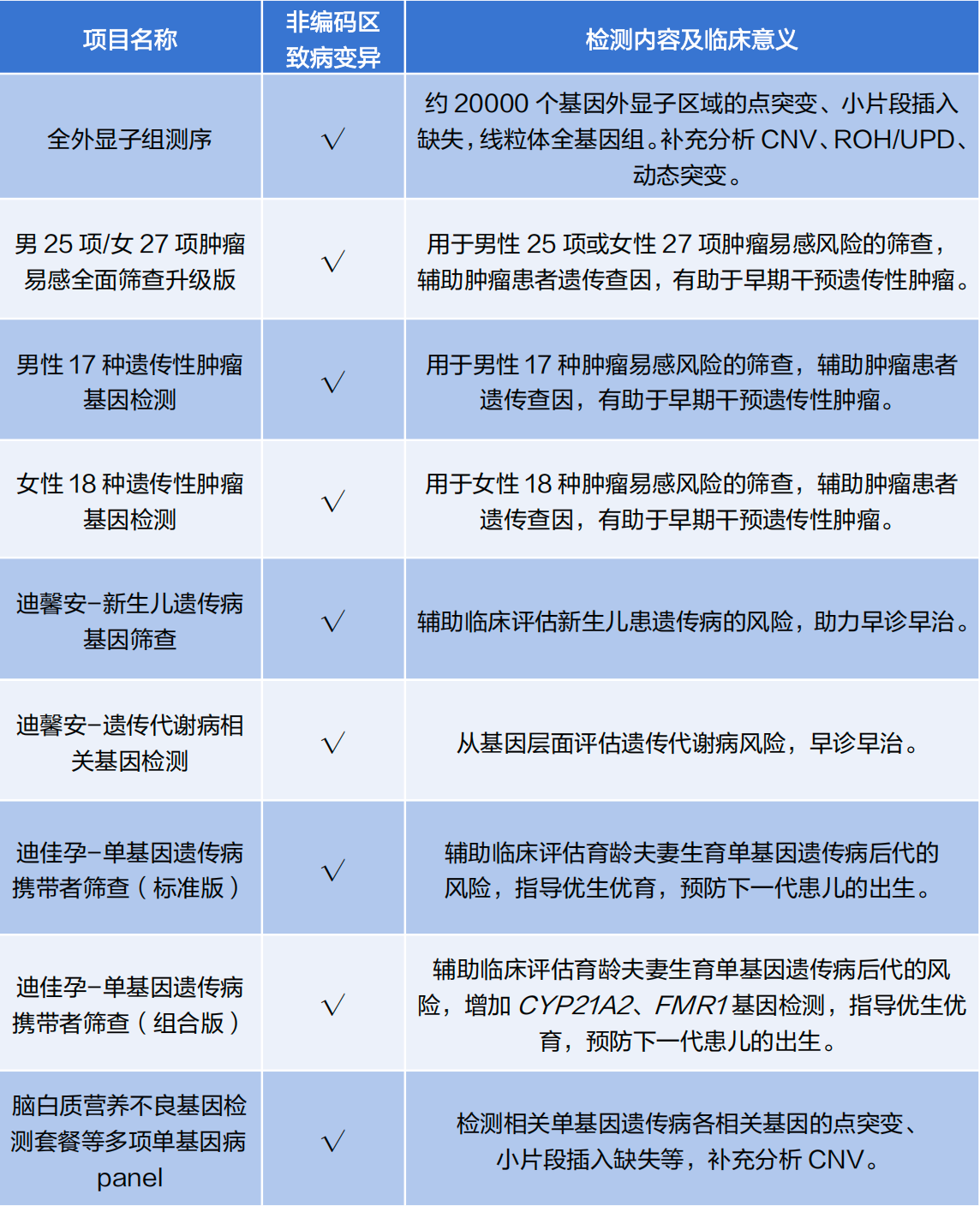
迪安诊断涉及非编码区致病变异的检测项目
参考文献
1. French JD, Edwards SL. The Role of Noncoding Variants in Heritable Disease. Trends Genet. 2020 Nov;36(11):880-891. Epub 2020 Jul 31)
2. Turner TN, Hormozdiari F, Duyzend MH,et al. Genome Sequencing of Autism-Affected Families Reveals Disruption of Putative Noncoding Regulatory DNA. Am J Hum Genet. 2016 Jan 7;98(1):58-74. Epub 2015 Dec 31
3. Carss KJ, Arno G, Erwood M, Stephens J, et al. Comprehensive Rare Variant Analysis via Whole-Genome Sequencing to Determine the Molecular Pathology of Inherited Retinal Disease. Am J Hum Genet. 2017 Jan 5;100(1):75-90. Epub 2016 Dec 29
4. Bertoli-Avella AM, Beetz C, Ameziane N, Rocha ME, et al. Successful application of genome sequencing in a diagnostic setting: 1007 index cases from a clinically heterogeneous cohort. Eur J Hum Genet. 2021 Jan;29(1):141-153. Epub 2020 Aug 28
5. Lord J, Oquendo CJ, Wai HA, et al. Noncoding variants are a rare cause of recessive developmental disorders in trans with coding variants. Genet Med. 2024 Dec;26(12):101249. Epub 2024 Sep 3





